Grupo do ICB descobre mecanismos genéticos relacionados à resistência de bactérias
Estudo tomou como base cepa da ‘E. coli’ isolada na UFMG em 1974 e que serve, desde então, sobretudo a atividades didáticas
Dados obtidos por grupo de pesquisadores do Instituto de Ciências Biológicas (ICB) da UFMG, em estudos com a cepa BH100 da bactéria Escherichia coli uropatogênica (Upec), revelam mecanismos por meio dos quais elementos genéticos móveis constituem fatores de disseminação de genes de resistência a drogas antimicrobianas. Os resultados estão publicados na edição de janeiro da revista Frontiers in Microbiology.
A cepa BH100 de E. coli foi isolada, em 1974, do trato urinário de uma brasileira pelo grupo do professor Edmar Chartone, da UFMG. Nos estudos de caracterização da bactéria, observou-se que a BH100 carrega dois plasmídeos (pBH100 e pAp) com propriedades conjugativas, o que significa que eles podem ser transferidos para outras cepas de E. coli. Além disso, o microrganismo era resistente a diversas drogas antimicrobianas de uso clínico e também ao mercúrio. A partir da década de 1980, variantes da BH100 foram obtidas por meio da eliminação dos plasmídeos e têm sido cultivadas em laboratório.
“Esse cenário singular foi condição favorável para a nossa investigação da atividade dos fatores que contribuem na mobilização de genes de resistência. Isso foi feito com a observação de rearranjos genômicos nas variantes que ainda continham os plasmídeos, na comparação com as variantes que haviam perdido esses elementos”, afirma o primeiro autor do artigo, Rodrigo Dias de Oliveira Carvalho, que faz estágio pós-doutoral no Laboratório de Genética Celular e Molecular do ICB, liderado pelo professor Vasco Azevedo. Posteriormente, conta o pesquisador, os genomas completos das variantes da BH100 foram sequenciados na Universidade de Göttingen, na Alemanha, por meio da plataforma de Sequenciamento de Nova Geração.
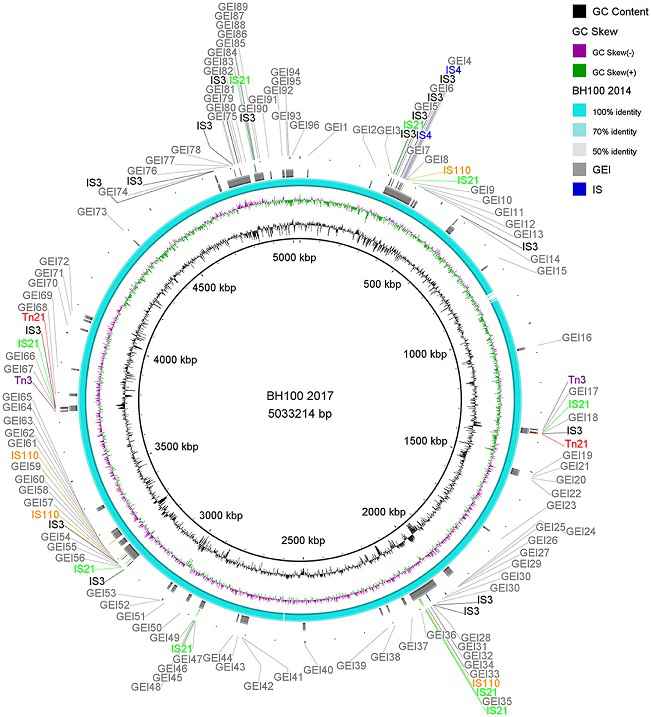
O estudo genômico – que incluiu as subcepas BH100 MG2014, BH100 MG2017, BH100L MG2017 e BH100N MG2017 – lançou mão de uma série de análises bioinformáticas conduzidas por Rodrigo Carvalho, incluindo controle de qualidade dos dados, montagem das sequências de DNA dos cromossomos e dos plasmídeos e predição de fatores genéticos relacionados à resistência e à virulência bacteriana.
Ilhas genômicas
No artigo recém-publicado, os autores explicam que Escherichia coli são bactérias encontradas naturalmente no trato intestinal de várias espécies animais e de humanos, onde algumas cepas podem coexistir com o hospedeiro em uma relação simbiótica, enquanto outras podem causar doenças. Um subconjunto de cepas de E. coli é capaz de causar doença extraintestinal, incluindo infecção do trato urinário (ITU). O patótipo uropatogênico Upec é responsável por 90% dos casos globais. Nesse contexto, as cepas multirresistentes vem sendo consideradas um grande gargalo para o controle efetivo de infecções, e o abuso de antibióticos tem sido crucial para a seleção de cepas com capacidade de adquirir genes de resistência a medicamentos por meio de mecanismos de transferência horizontal.

De acordo com Rodrigo Carvalho, os dados obtidos na pesquisa sugerem a ocorrência de eventos de recombinação entre os plasmídeos pBH100 e pAp e também entre eles e o cromossomo da E. coli BH100. “Em ambos os casos, foram identificados os elementos IS3 e IS21, que codificam genes de transposição e podem ser responsáveis pelos fenômenos de mobilização de genes de resistência a antibióticos na região recombinada”, afirma o pesquisador.
Carvalho acrescenta que esse resultado indica a possibilidade de que tais elementos tenham papel relevante para a adaptação de bactérias multirresistentes, por meio da formação de ilhas genômicas de resistência, regiões do cromossomo bacteriano que conferem plasticidade genômica ao patógeno. “Isso significa que essas ilhas atuam na transferência de segmentos de material genético que conferem a outras bactérias propriedades de resistência a drogas e capacidade de provocar doenças”, afirma.
Segundo o pesquisador, muitos dos fatores genéticos relacionados à resistência antimicrobiana encontrados no genoma da E. coli BH100 também foram detectados em outras cepas de Upec mais recentes e até mesmo em outras espécies: “Isso leva a concluir que a BH100 pode ter contribuído para a aquisição de resistência por parte dessas bactérias". É ponto pacífico que as bactérias multirresistentes são um dos grandes problemas de saúde pública. Rodrigo Carvalho enfatiza que, ao prover novas informações para a compreensão dos mecanismos relacionados à aquisição de resistência a drogas antimicrobianas pelos patógenos, o trabalho recém-publicado contribui para a elaboração de estratégias e medidas de controle mais eficazes de infecções hospitalares.
O trabalho também teve participação de pesquisadores das universidades federais de Pernambuco (UFPE), do Pará (UFPA), do Triângulo Mineiro (UFTM) e de instituições da Rússia, da Alemanha, da Índia e dos Estados Unidos. Essas são algumas das integrantes da Rede de Ciências Ômicas (Recom), formada por grupos de pesquisa que trabalham com a bioinformática para lidar com grandes volumes de dados biológicos. Um dos objetivos da rede, coordenada pela UFMG, pela UFTM, pela UFPA e pela Universidade Federal do Vale São Francisco (Univasf), é estimular a formação em bioinformática em outras universidades.
Artigo: Genomic characterization of multidrug-resistant Escherichia coli BH100 sub-strains
Autores: Rodrigo Carvalho, Flavia Aburjaile, Marcus Canario, Andréa M.A. Nascimento, Edmar Chartone-Souza, Luis de Jesus, Andrey A. Zamyatnin Jr., Bertram Brenig, Debmalya Barh, Preetam Ghosh, Aristoteles Goes-Neto, Henrique C.P. Figueiredo, Siomar Soares, Rommel Ramos, Anne Pinto e Vasco Azevedo
Publicação: Frontiers in Microbiology, em janeiro de 2021